Data-Mining
Cours-TP sur la validation des méthodes d'apprentissage
Contents
Description du TP
Le but de ce TP est de comprendre et mettre en oeuvre des méthodes de validation utlisée lors de la calibration de méthodes d'apprentissage.
Nous allons comparer les approches naïves aux approches de validation croisée et de leave-one-out.
function CoursTP_VC_ROC()
clear all; close all;
Les données
Nous allons utiliser des données synthétiques, aussi appelées données jouet, dans un but d'illustration.
nombre de points en apprentissage
N = 50;
nombre de points en test
Ntest = 1000;
génération et mélange des données
x1 = [0.3*randn(round(N/2),2)+0.3;0.35*randn(round(N/2),2)-0.2]; y1 = [ones(round(N/2),1);-ones(round(N/2),1)]; temp = randperm(length(y1)); x1 = x1(temp,:); y1 = y1(temp); xtest1 = [0.3*randn(round(Ntest/2),2)+0.3;0.35*randn(round(Ntest/2),2)-0.2]; ytest1 = [ones(round(Ntest/2),1);-ones(round(Ntest/2),1)];
On a besoin de savoir quelle est la première classe rencontrée par l'algorithme d'apprentissage
c1 = y1(1); c2 = -c1;
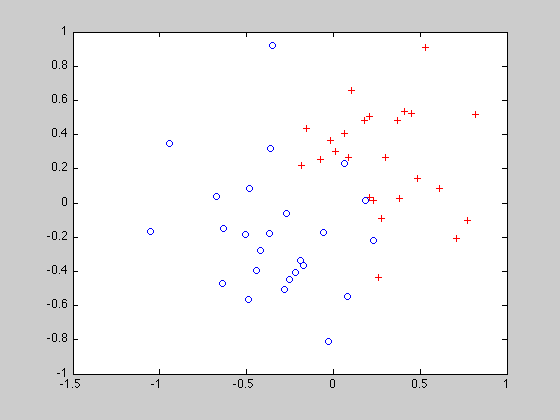
Visualisation
figure; plot(x1(y1==1,1),x1(y1==1,2),'r+'); hold on; plot(x1(y1==-1,1),x1(y1==-1,2),'bo');
SVM
La méthode d'apprentissage que nous allons utiliser pour ce TP est appelée SVM (Séparateurs à Vaste Marge). Cette méthode
permet de faire de la discrimination et requière le réglage de plusieurs paramètres. Sans rentrer dans les détails, nous allons
ici nous intéresser au réglage de 2 paramètres, nommés respectivement 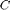 et
et  .
.
La toolbox est disponible là et les instruction pour pouvoir l'utiliser sont là Tout d'abord, il faut ajouter le chemin jusqu'aux exécutables du SVM
addpath('../Toolbox/libsvm-mat-2.88-1/');
Paramètres
le paramètre 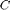 prend une valeur réelle strictement positive. Habituellement, l'ordre de grandeur varie entre 1 et 10000. le paramètre
prend une valeur réelle strictement positive. Habituellement, l'ordre de grandeur varie entre 1 et 10000. le paramètre  est également un réel strictement positif et est lié à une notion d'écart-type :
est également un réel strictement positif et est lié à une notion d'écart-type :  .
.
Prennons deux valeurs, correspondant d'une part à une faible tolérance aux erreurs d'apprentissage (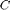 grand) et d'autre part à une très faible largeur de bande (
grand) et d'autre part à une très faible largeur de bande ( grand), soit une faible influence de chaque point dans la solution.
grand), soit une faible influence de chaque point dans la solution.
C = 1000; gamma = 100; param = ['-b 1 -c ', num2str(C), ' -g ', num2str(gamma)];
Lançons l'apprentissage
model = svmtrain(y1, x1, param);
Testons la performance du modèle sur les données d'apprentissage
disp('Performance en apprentissage');
[p] = svmpredict(y1, x1, model);
Performance en apprentissage Model supports probability estimates, but disabled in predicton. Accuracy = 100% (50/50) (classification)
Puis testons sur des données de test et affichons la décision rendue par le SVM sur tout l'espace.
disp('Performance en test'); [predict_label, accuracy, proba] = svmpredict(ytest1, xtest1, model, '-b 1');
Performance en test Accuracy = 74.5% (745/1000) (classification)
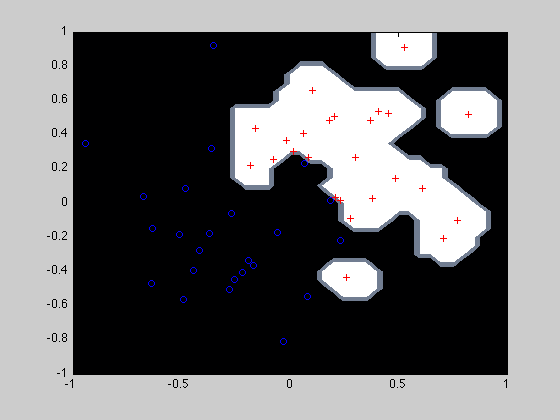
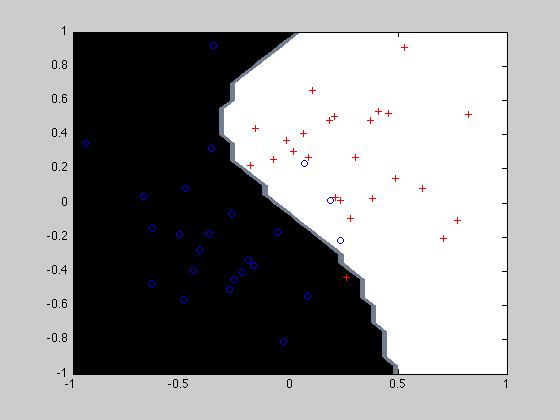
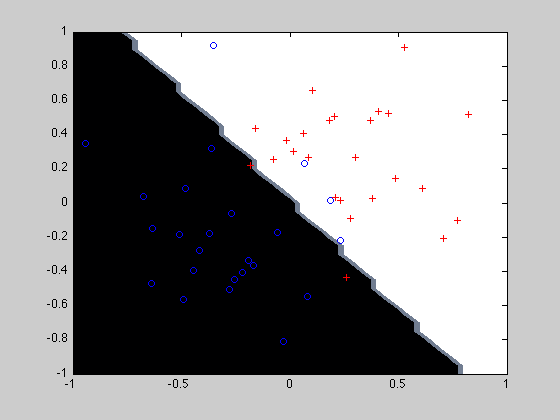
Affichage du SVM Le code de l'affichage se trouve là
afficherSVM(model, x1,y1);
------------------------------------ Les valeurs affichées ci-dessous n'ont pas de sens car les étiquettes sont absentes... Model supports probability estimates, but disabled in predicton. Accuracy = 0% (0/1681) (classification) ------------------------------------
Affichage de la matrice de confusion
Le code de la matrice de confusion se trouve là
MCBase = matriceConfusion(ytest1,predict_label,c1,c2)
MCBase =
310 190
65 435
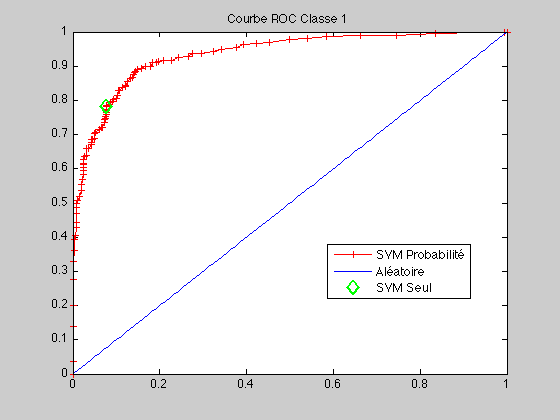
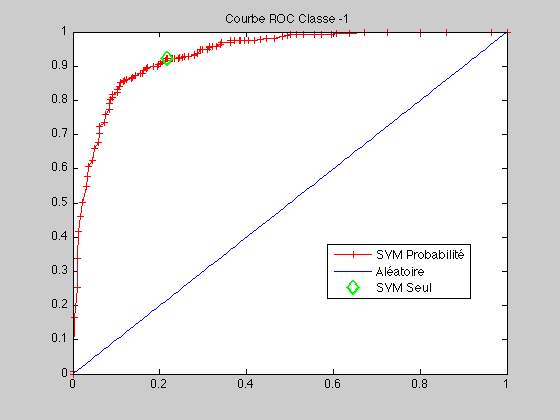
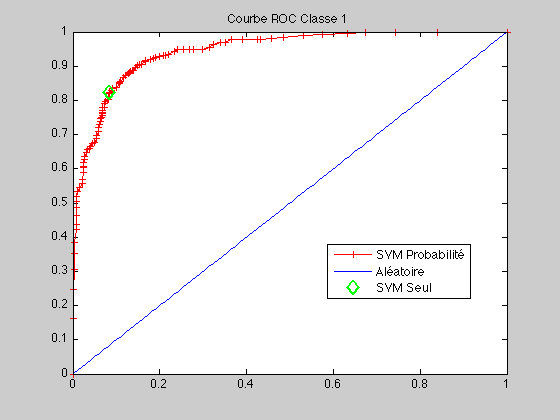
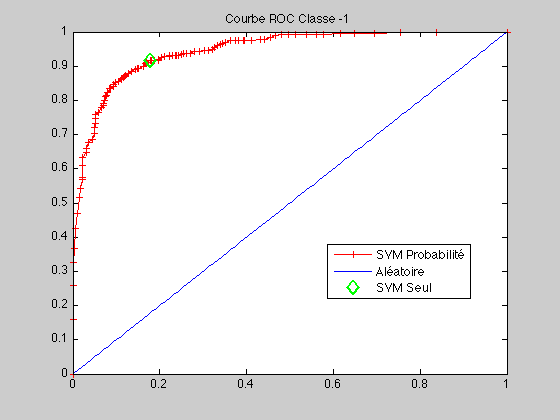
Etudions la courbe ROC Le code de la courbe ROC se trouve là
courbeROC(c1,c2,ytest1,proba,predict_label)
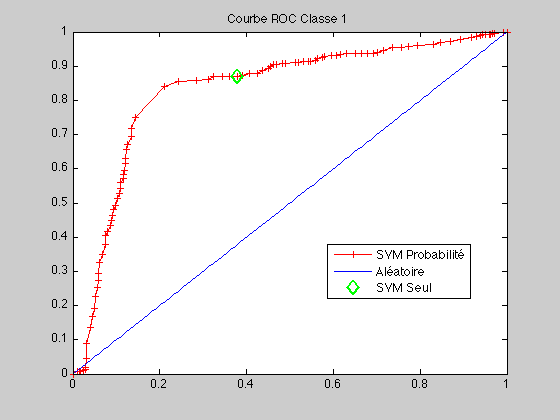
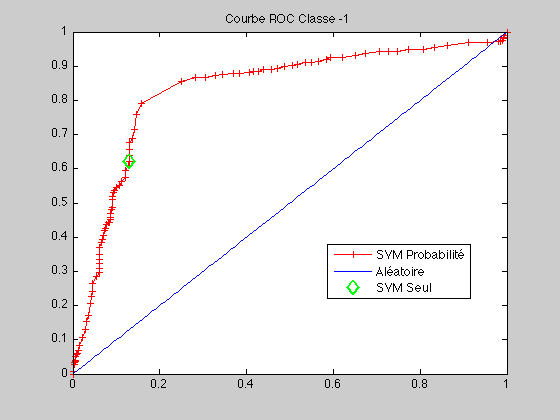
Prennons deux autres valeurs
C = 10; gamma = 1; param = ['-b 1 -c ', num2str(C), ' -g ', num2str(gamma)]; model = svmtrain(y1, x1, param); disp('Performance en apprentissage'); [p] = svmpredict(y1, x1, model); disp('Performance en test'); [predict_label, accuracy, proba] = svmpredict(ytest1, xtest1, model, '-b 1'); afficherSVM(model, x1,y1); MCBase = matriceConfusion(ytest1,predict_label,c1,c2) courbeROC(c1,c2,ytest1,proba,predict_label);
Performance en apprentissage Model supports probability estimates, but disabled in predicton. Accuracy = 92% (46/50) (classification) Performance en test Accuracy = 85.2% (852/1000) (classification) ------------------------------------ Les valeurs affichées ci-dessous n'ont pas de sens car les étiquettes sont absentes... Model supports probability estimates, but disabled in predicton. Accuracy = 0% (0/1681) (classification) ------------------------------------ MCBase = 461 39 109 391
Validation croisée
La validation croisée permet de vérifier la pertinence d'un ensemble de tuples de paramètres sans utiliser les données de
test, en utilisant un découpage de la base d'apprentissage en  morceaux. Chaque morceau sert une fois de jeu de validation pendant que le complément sert de base d'apprentissage. La performance
d'un tuple de paramètre est évalué par moyennage (voir code de la validation croisée qui se trouve là)
morceaux. Chaque morceau sert une fois de jeu de validation pendant que le complément sert de base d'apprentissage. La performance
d'un tuple de paramètre est évalué par moyennage (voir code de la validation croisée qui se trouve là)
Choix des paramètres à tester
Choisir le nombre de découpages pour la validation croisée, la liste de valeurs de paramètres à tester, et effectuer cette validation.
nbFolders = 3; C = [1 10 100 1000 10000]; bandwidth = [0.01 0.1 1 10]; [I,J] = crossValidationClassBin(x1,y1,nbFolders,C,bandwidth);
Accuracy = 41.1765% (7/17) (classification) Accuracy = 94.1176% (16/17) (classification) Accuracy = 94.1176% (16/17) (classification) Accuracy = 88.2353% (15/17) (classification) Accuracy = 94.1176% (16/17) (classification) Accuracy = 82.3529% (14/17) (classification) Accuracy = 94.1176% (16/17) (classification) Accuracy = 94.1176% (16/17) (classification) Accuracy = 82.3529% (14/17) (classification) Accuracy = 88.2353% (15/17) (classification) Accuracy = 88.2353% (15/17) (classification) Accuracy = 88.2353% (15/17) (classification) Accuracy = 82.3529% (14/17) (classification) Accuracy = 88.2353% (15/17) (classification) Accuracy = 88.2353% (15/17) (classification) Accuracy = 88.2353% (15/17) (classification) Accuracy = 82.3529% (14/17) (classification) Accuracy = 82.3529% (14/17) (classification) Accuracy = 88.2353% (15/17) (classification) Accuracy = 88.2353% (15/17) (classification) Accuracy = 47.0588% (8/17) (classification) Accuracy = 76.4706% (13/17) (classification) Accuracy = 76.4706% (13/17) (classification) Accuracy = 82.3529% (14/17) (classification) Accuracy = 76.4706% (13/17) (classification) Accuracy = 76.4706% (13/17) (classification) Accuracy = 82.3529% (14/17) (classification) Accuracy = 70.5882% (12/17) (classification) Accuracy = 76.4706% (13/17) (classification) Accuracy = 82.3529% (14/17) (classification) Accuracy = 82.3529% (14/17) (classification) Accuracy = 70.5882% (12/17) (classification) Accuracy = 82.3529% (14/17) (classification) Accuracy = 76.4706% (13/17) (classification) Accuracy = 70.5882% (12/17) (classification) Accuracy = 70.5882% (12/17) (classification) Accuracy = 76.4706% (13/17) (classification) Accuracy = 64.7059% (11/17) (classification) Accuracy = 70.5882% (12/17) (classification) Accuracy = 70.5882% (12/17) (classification) Accuracy = 43.75% (7/16) (classification) Accuracy = 93.75% (15/16) (classification) Accuracy = 87.5% (14/16) (classification) Accuracy = 87.5% (14/16) (classification) Accuracy = 93.75% (15/16) (classification) Accuracy = 93.75% (15/16) (classification) Accuracy = 87.5% (14/16) (classification) Accuracy = 75% (12/16) (classification) Accuracy = 93.75% (15/16) (classification) Accuracy = 93.75% (15/16) (classification) Accuracy = 87.5% (14/16) (classification) Accuracy = 75% (12/16) (classification) Accuracy = 93.75% (15/16) (classification) Accuracy = 93.75% (15/16) (classification) Accuracy = 81.25% (13/16) (classification) Accuracy = 75% (12/16) (classification) Accuracy = 93.75% (15/16) (classification) Accuracy = 87.5% (14/16) (classification) Accuracy = 81.25% (13/16) (classification) Accuracy = 75% (12/16) (classification)
SVM
Lancer l'apprentissage final avec les paramètres issus de la validation croiée
bestGs = bandwidth(J(1)) bestCs = C(I(1)) param = ['-b 1 -c ', num2str(bestCs), ' -g ', num2str(bestGs)]; model = svmtrain(y1, x1, param); disp('Performance en apprentissage'); [p] = svmpredict(y1, x1, model); disp('Performance en test'); [predict_label, accuracy, proba] = svmpredict(ytest1, xtest1, model, '-b 1'); afficherSVM(model, x1,y1); MCBase = matriceConfusion(ytest1,predict_label,c1,c2) courbeROC(c1,c2,ytest1,proba,predict_label);
bestGs =
0.0100
bestCs =
10
Performance en apprentissage
Model supports probability estimates, but disabled in predicton.
Accuracy = 88% (44/50) (classification)
Performance en test
Accuracy = 86.9% (869/1000) (classification)
------------------------------------
Les valeurs affichées ci-dessous n'ont pas de sens car les étiquettes sont absentes...
Model supports probability estimates, but disabled in predicton.
Accuracy = 0% (0/1681) (classification)
------------------------------------
MCBase =
458 42
89 411
end