Validation Croisée
(Voir cours)
Contents
Input
- trainvec matrice des données d'apprentissage
- trainlab vecteur des étiquettes d'apprentissage
- nbFolders nombre de groupes pour le découpage
- Cs liste des valeurs du paramètre
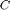
- Ks liste des valeurs du paramètre

Output
- I meilleur(s) paramètre(s)
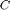 (plusieurs si performance équivalente en moyenne)
(plusieurs si performance équivalente en moyenne)
- J meilleur(s) paramètre(s)
 (plusieurs si performance équivalente en moyenne)
(plusieurs si performance équivalente en moyenne)
function [I,J] = crossValidationClassBin(trainvec,trainlab,nbFolders,Cs,Ks)
if nargin==5 precision = zeros(length(Cs),length(Ks)); else error('Pb arguments : 5 arguments attendus'); end
Précalculs
On calcule un nouvel ordre des éléments de la base. Cela permet d'éviter une sous base consitituée d'éléments de la même classe.
rn = randperm(length(trainlab));
Puis on calcul la taille des sous bases
S = ceil(length(trainlab)/nbFolders);
Exécution et comparaison
for N = 1:nbFolders
On sélectionne pour base de validation le sous-groupe N
selected = rn((S*(N-1))+1:min(end,N*S));
TTV = trainvec(selected,:);
TTL = trainlab(selected);
Le reste des données consistue la base d'apprentissage
notselected = setdiff(rn,selected);
TV = trainvec(notselected,:);
TL = trainlab(notselected);
On lance l'apprentissage et le test pour chaque couple de paramètres sur les bases courantes
for i=1:length(Cs) for j=1:length(Ks) param = ['-s 0 -c ', num2str(Cs(i)), ' -g ', num2str(Ks(j))]; model = svmtrain(TL,TV, param); [predict_label, accuracy] = svmpredict(TTL,TTV, model); precision(i,j) = precision(i,j)+accuracy(1); end end
end
Moyennage
On calcule la moyenne des performances pour chaque couple de paramètres
precision = precision./nbFolders;
On récupère le ou les couples donnant la meilleurs performance en moyenne
A = find(precision == max(max(precision))); [I,J] = ind2sub(size(precision),A);
end