TP 2 - SVM
Le but de ce TP est de mettre en oeuvre les SVM sur des données synthétiques afin d'observer le comportement en fonction des paramètres et des données.
Contents
Pour commencer
clear all; close all; % créer les données [trainvec,trainlab,testvec,testlab] = createData (500,1000,2,0.2); % choisir des parametres - la validation croisée serait bien ici! C = 1000; G = 1; % ajouter les chemins vers la toolbox SVM addpath('./toolbox/libsvm'); % lancer le SVM param = ['-b 1 -c ', num2str(C), ' -g ', num2str(G)]; model = svmtrain(trainlab,trainvec, param); disp('Performance en apprentissage'); [p] = svmpredict(trainlab, trainvec, model); disp('Performance en test'); [predict_label, accuracy, proba] = svmpredict(testlab, testvec, model, '-b 1'); % afficher quand les données sont en 2D afficherSVM(model, trainvec,trainlab); % regarder la matrice de confusion MCBase = matriceConfusion(testlab,predict_label,1,-1)
Performance en apprentissage Model supports probability estimates, but disabled in predicton. Accuracy = 86.4919% (429/496) (classification) Performance en test Accuracy = 75.8065% (752/992) (classification) ------------------------------------ Les valeurs affichées ci-dessous n'ont pas de sens car les étiquettes sont absentes... Model supports probability estimates, but disabled in predicton. Accuracy = 0% (0/6561) (classification) ------------------------------------ MCBase = 377 119 121 375
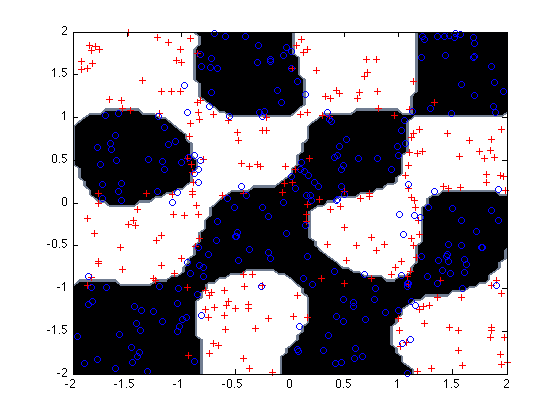
Votre contribution
Les éléments à observer sont les suivants
- Sur un problème donné, lancer le SVM en faisant varier le paramètre C et observer son effet (choisir un damier avec mélange)
- Sur un problème donné, lancer le SVM avec différents noyaux (gaussien (rbf),lineaire, polynomial)
- Mettre en place la validation croisée pour un noyau gaussien (libSVM propose un mode validation croisée, qui pour un réglage retourne la performance en validation...)