TP 4 : Les cartes de Kohonen
Contents
Outils
Lors de ce TP , nous utiliserons une boîte à outils dédiée aux Cartes de Kohonen (disponible ici).
clear all; close all; addpath('./Toolbox/somtoolbox');
Présentation des cartes de Kohonen
Les cartes de Kohonen, aussi appelées carte auto-organisatrices (Self Organizing Maps : SOM), sont une forme de réseau de neurones. Dans ce type de réseau, les neurones sont connectés entre eux selon une notion de voisinage et non selon une notion de couche.
figure; som_cplane('hexa',[10 15],'none'); title('Grille SOM hexagonale : chaque neurone est connecté à 6 voisins'); figure; som_cplane('rect',[10 15],'none'); title('Grille SOM rectangulaire : chaque neurone est connecté à 4 voisins');


Extrait de la documentation de la toolbox SOM
- som_make - Create, initialize and train a SOM.
- som_randinit - Create and initialize a SOM.
- som_lininit - Create and initialize a SOM.
- som_seqtrain - Train a SOM.
- som_batchtrain - Train a SOM.
- som_bmus - Find best-matching units (BMUs).
- som_quality - Measure quality of SOM.
SELF-ORGANIZING MAP (SOM):
% * A self-organized map (SOM) is a "map" of the training data, % dense where there is a lot of data and thin where the data % density is low. % * The map constitutes of neurons located on a regular map grid. % * The lattice of the grid can be either hexagonal or rectangular. % * Each neuron (hexagon on the left, rectangle on the right) has an % associated prototype vector. After training, neighboring neurons % have similar prototype vectors. % * The SOM can be used for data visualization, clustering (or % classification), estimation and a variety of other purposes.
Initialisation et apprentissage d'une carte
Dans un premier temps, nous générons 600 points selon deux gaussiennes distinctes dans un espace en 2 dimension.
D = [0.3*randn(300,2)-0.5;0.1*randn(300,2)+0.4]; sData = som_data_struct(D,'name','gausians','comp_names',{'x','y'});
Les étiquettes peuvent être renseignées (toutefois les cartes SOM sont des méthodes non supervisées, c'est-à-dire que les étiquettes ne sont pas utilisées pour l'apprentisssage)
sData = som_label(sData,'add',[1:300]','1'); sData = som_label(sData,'add',[301:600]','2');
Choix des dimensions de la carte, ici en 2 dimensions (si la taille n'est pas définie, elle est choisie automatiquement en fonction des valeurs et vecteurs propres des données)
msize = [10 10];
Initialisation aléatoire des poids du réseau
sMap = som_randinit(D, 'msize', msize);
En fait, chaque neurone de la carte peut être envisagé selon deux points de vue : * dans l'espace de représentation, selon les coordonnées de la carte * dans l'espace des données, selon leur position par rapport à leur prototype.
figure; som_grid(sMap) title('Carte dans l''espace de représentation') figure; plot(D(:,1),D(:,2),'+r'), hold on som_grid(sMap,'Coord',sMap.codebook) title('Carte dans l''espace des données')
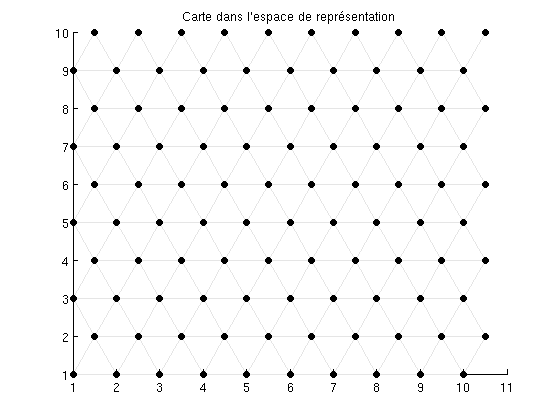
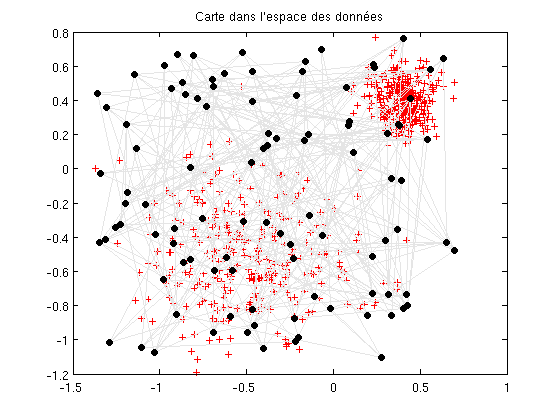
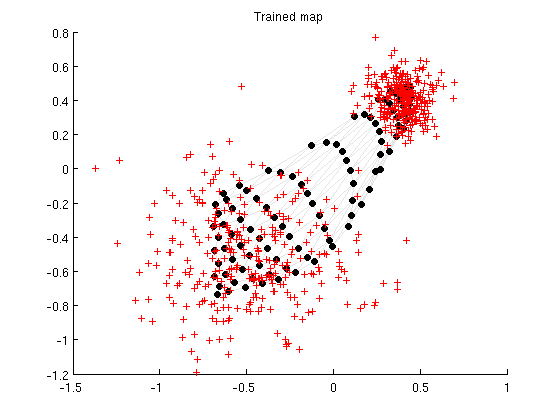
Dans les figures, les points noirs représentent la position de chaque neurone de la carte, les lignes grises montrent les connexions de voisinnage. Puisque la carte est initialisée aléatoirement, les neurones sont complètement désorganisés dans l'espace des données. Au cours de l'apprentissage, la carte s'organise en utilisant la ressemblence entre les données d'apprentissage. (tapez help som_septrain pour plus d'informations sur les options d'apprentissage)
sMap = som_seqtrain(sMap,D,'radius',[5 1],'trainlen',10); figure; som_grid(sMap,'Coord',sMap.codebook) hold on, plot(D(:,1),D(:,2),'+r') title('Trained map')
Training: 0/ 1 sTraining: 0/ 1 sTraining: 0/ 0 sTraining: 0/ 0 sTraining: 0/ 0 sTraining: 0/ 0 sTraining: 0/ 0 sTraining: 0/ 0 sTraining: 0/ 0 sTraining: 0/ 0 sTraining: 0/ 0 sTraining: 0/ 0 sTraining: 0/ 0 sTraining: 0/ 0 sTraining: 0/ 0 sTraining: 0/ 0 sTraining: 0/ 0 sTraining: 0/ 0 sTraining: 0/ 0 sTraining: 0/ 0 sTraining: 0/ 0 sTraining: 0/ 0 sTraining: 0/ 0 sTraining: 0/ 0 sTraining: 0/ 0 sTraining: 0/ 0 sTraining: 0/ 0 sTraining: 0/ 0 sTraining: 0/ 0 sTraining: 0/ 0 sTraining: 0/ 0 sTraining: 0/ 0 sTraining: 0/ 0 sTraining: 0/ 0 sTraining: 0/ 0 sTraining: 0/ 0 sTraining: 0/ 0 sTraining: 0/ 0 sTraining: 0/ 0 sTraining: 0/ 0 sTraining: 0/ 0 sTraining: 0/ 0 sTraining: 0/ 0 sTraining: 0/ 0 sTraining: 0/ 0 sTraining: 0/ 0 sTraining: 0/ 0 sTraining: 0/ 0 sTraining: 0/ 0 sTraining: 0/ 0 sTraining: 0/ 0 sTraining: 0/ 0 sTraining: 0/ 0 sTraining: 0/ 0 sTraining: 0/ 0 sTraining: 0/ 0 sTraining: 0/ 0 sTraining: 0/ 0 sTraining: 0/ 0 sTraining: 0/ 0 s
Déroulement de l'apprentissage
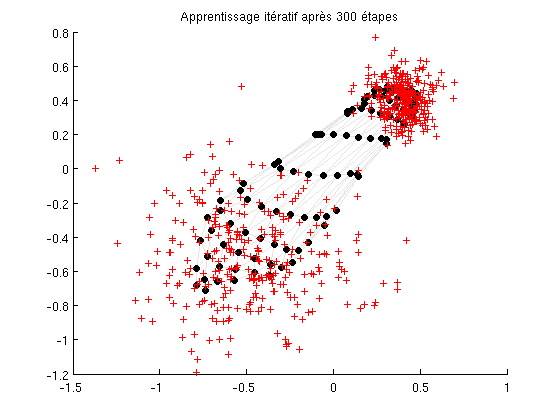
L'exemple ci-après montre comment la carte se déplie selon les données au cours de l'apprentissage. Pour que la visualisation soit meilleure, on initialise la carte loin des données de départ.
sMap = som_randinit(D,'msize',msize); sMap.codebook = sMap.codebook + 3; figure; som_grid(sMap,'Coord',sMap.codebook) hold on, plot(D(:,1),D(:,2),'+r'), hold off title('Data and original map')
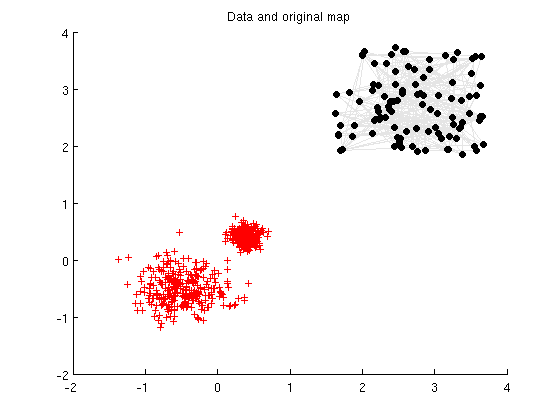
L'apprentissage se fait selon 2 principes : * la compétition : le prototype qui ressemble le plus à une donnée d'apprentissage est modifié de manière à lui ressembler encore plus. Ainsi chaque neurone de la carte se spécialise et est de plus en plus représentatif des données. * la coopération : les voisins du neurone dont le prototype est modifié sont également modifiés, dans une moindre mesure, de manière à se rapprocher de l'exemple. C'est ainsi que la carte s'auto-organise.
figure; o = ones(5,1); r = (1-[1:60]/60); for i=1:60, sMap = som_seqtrain(sMap,D,'tracking',0,... 'trainlen',5,'samples',... 'alpha',0.1*o,'radius',(4*r(i)+1)*o); som_grid(sMap,'Coord',sMap.codebook) hold on, plot(D(:,1),D(:,2),'+r'), hold off title(sprintf('%d/300 training steps',5*i)) drawnow end title('Apprentissage itératif après 300 étapes')
Comparaison avec les étiquettes
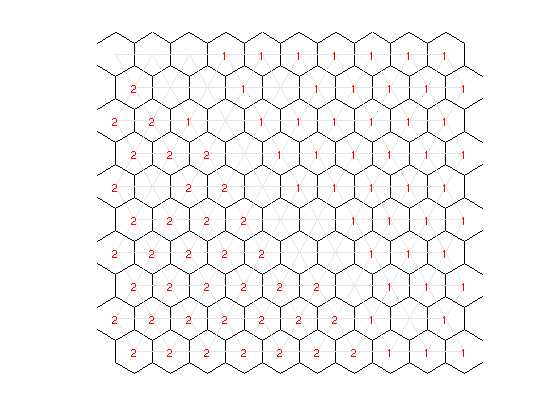
Il est possible de visualiser la performance de la carte en affichant, pour chaque neurone, l'étiquette la plus représentée par son prototype.
sMap = som_autolabel(sMap,sData,'vote'); figure; som_cplane(sMap,'none'); hold on; som_grid(sMap,'Label',sMap.labels,'Labelsize',8,... 'Marker','none','Labelcolor','r');
Utilisation de la carte pour faire de la projection de données
Dans cette partie, nous allons utiliser des données en dimension 10, réparties selon 5 classes, chacune générée selon une gaussienne particulière.
D = [0.4*randn(100,10)-ones(100,1)*randn(1,10);0.4*randn(100,10)+ones(100,1)*randn(1,10);... 0.4*randn(100,10)+ones(100,1)*randn(1,10);0.6*randn(100,10)+ones(100,1)*randn(1,10);... 0.6*randn(100,10)+ones(100,1)*randn(1,10)]; sData = som_data_struct(D,'name','gausians'); sData = som_label(sData,'add',[1:100]','1'); sData = som_label(sData,'add',[101:200]','2'); sData = som_label(sData,'add',[201:300]','3'); sData = som_label(sData,'add',[301:400]','4'); sData = som_label(sData,'add',[401:500]','5');
La carte est initialisée aléatoirement, le choix de la taille de la grille est automatique.
sMap = som_randinit(D); sMap = som_seqtrain(sMap,D,'radius',[2 0.5],'trainlen',20);
Training: 0/ 1 sTraining: 0/ 1 sTraining: 0/ 1 sTraining: 0/ 1 sTraining: 0/ 1 sTraining: 0/ 1 sTraining: 0/ 1 sTraining: 0/ 1 sTraining: 0/ 1 sTraining: 0/ 1 sTraining: 0/ 1 sTraining: 0/ 1 sTraining: 0/ 1 sTraining: 0/ 1 sTraining: 0/ 1 sTraining: 0/ 1 sTraining: 0/ 1 sTraining: 0/ 1 sTraining: 0/ 1 sTraining: 0/ 1 sTraining: 0/ 1 sTraining: 0/ 1 sTraining: 0/ 1 sTraining: 0/ 1 sTraining: 0/ 1 sTraining: 0/ 1 sTraining: 0/ 1 sTraining: 0/ 1 sTraining: 0/ 1 sTraining: 0/ 1 sTraining: 0/ 1 sTraining: 0/ 1 sTraining: 0/ 1 sTraining: 0/ 1 sTraining: 0/ 1 sTraining: 0/ 1 sTraining: 0/ 1 sTraining: 0/ 1 sTraining: 0/ 1 sTraining: 0/ 1 sTraining: 0/ 1 sTraining: 0/ 1 sTraining: 0/ 1 sTraining: 0/ 1 sTraining: 0/ 1 sTraining: 0/ 1 sTraining: 0/ 1 sTraining: 0/ 1 sTraining: 0/ 1 sTraining: 0/ 1 sTraining: 0/ 1 sTraining: 0/ 1 sTraining: 0/ 1 sTraining: 0/ 1 sTraining: 0/ 1 sTraining: 0/ 1 sTraining: 0/ 1 sTraining: 0/ 1 sTraining: 0/ 1 sTraining: 0/ 1 sTraining: 0/ 1 sTraining: 0/ 1 sTraining: 0/ 1 sTraining: 0/ 1 sTraining: 0/ 1 sTraining: 0/ 1 sTraining: 0/ 1 sTraining: 0/ 1 sTraining: 0/ 1 sTraining: 0/ 1 sTraining: 0/ 1 sTraining: 0/ 1 sTraining: 0/ 1 sTraining: 0/ 1 sTraining: 0/ 1 sTraining: 0/ 1 sTraining: 0/ 1 sTraining: 0/ 1 sTraining: 0/ 1 sTraining: 0/ 1 sTraining: 0/ 1 sTraining: 0/ 1 sTraining: 0/ 1 sTraining: 0/ 1 sTraining: 1/ 1 sTraining: 1/ 1 sTraining: 1/ 1 sTraining: 1/ 1 sTraining: 1/ 1 sTraining: 1/ 1 sTraining: 1/ 1 sTraining: 1/ 1 sTraining: 1/ 1 sTraining: 1/ 1 sTraining: 1/ 1 sTraining: 1/ 1 sTraining: 1/ 1 sTraining: 1/ 1 sTraining: 1/ 1 sTraining: 1/ 1 s
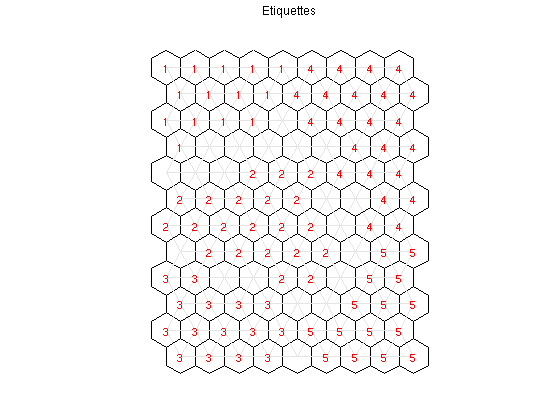
La carte étant en 2 dimension, nous avons automatiquement une réduction de dimension de données...
figure; som_cplane(sMap,'none') sMap = som_autolabel(sMap,sData,'vote'); hold on som_grid(sMap,'Label',sMap.labels,'Labelsize',8,... 'Marker','none','Labelcolor','r'); hold off title('Etiquettes')
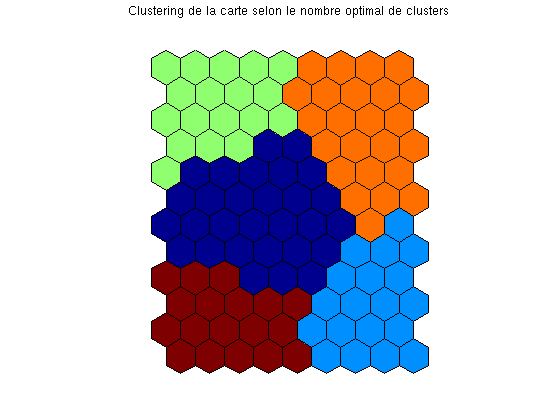
Recherche de cluster.
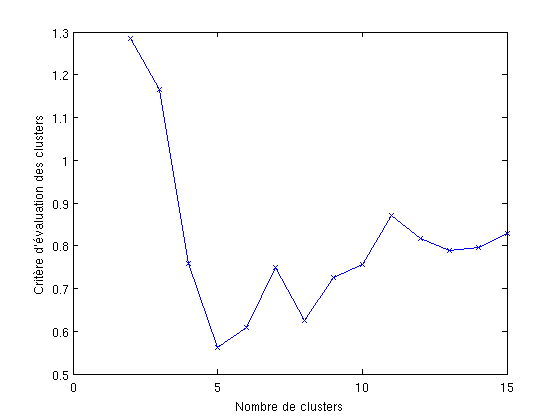
Dans l'apprentissage non supervisé, on recherche généralement des ensembles de données se ressemblant tout en étant différents des autres ensembles, c'est ce que l'on appelle des clusters. Ici on utilise l'algorithme des k-moyennes pour trouver des clusters sur la carte en 2D. On va rechercher au plus 15 cluster, une mesure de qualité des cluster permettra de déterminer automatiquement combien vont être retenus.
figure; [c,p,err,ind] = kmeans_clusters(sMap, 15); plot(1:length(ind),ind,'x-') xlabel('Nombre de clusters') ylabel('Critère d''évaluation des clusters') [dummy,i] = min(ind) cl = p{i}; %subplot(1,3,2) %som_cplane(sMap,Code,Dm) %title('Carte représentant la fréquence d''utilisation de chaque neurone') figure som_cplane(sMap,cl) title('Clustering de la carte selon le nombre optimal de clusters')
dummy =
0.5620
i =
5
Travail à réaliser
Reprennez les données USPS (les 10 classes) et construisez une carte de Kohonen dédiée. Visualisez la répartition des étiquettes, essayez de faire du clustering, regardez les effets de la taille de la carte (automatique, arbitrairement grande ou petite...). Le compte rendu contiendra la figure représentant les étiquettes d'apprentissage sur la carte. Il fera aussi figurer le taux de bonne classification sur les données de test.
Code à utiliser pour le test...
ypred1 = zeros(size(test_labels));
for i=1:length(test_labels)
y1 = som_hits(sMap,test_patterns(:,i)');
ypred1(i) = str2double(sMap.labels{y1==max(y1)});
end
testErr1 = length(find(ypred1==test_labels))/length(test_labels)somUsps;
Training: 0/ 4 sTraining: 0/ 5 sTraining: 1/ 5 sTraining: 1/ 5 sTraining: 1/ 5 sTraining: 2/ 5 sTraining: 2/ 5 sTraining: 2/ 5 sTraining: 2/ 5 sTraining: 2/ 5 sTraining: 3/ 5 sTraining: 3/ 5 sTraining: 3/ 5 sTraining: 3/ 5 sTraining: 4/ 5 sTraining: 4/ 5 sTraining: 4/ 5 sTraining: 4/ 5 sTraining: 5/ 5 sTraining: 5/ 5 s
dummy =
1.4074
i =
15
bonneClassification =
0.8984

